Análise COVID-19 em SP - Dados do Hospital Albert Einstein
14 minuto(s) de leitura
import pandas as pd
import matplotlib.pyplot as plt
import numpy as np
pd.set_option('display.max_columns', 200)
df = pd.read_excel('dataset.xlsx')
df.head()
|
Patient ID |
Patient age quantile |
SARS-Cov-2 exam result |
Patient addmited to regular ward (1=yes, 0=no) |
Patient addmited to semi-intensive unit (1=yes, 0=no) |
Patient addmited to intensive care unit (1=yes, 0=no) |
Hematocrit |
Hemoglobin |
Platelets |
Mean platelet volume |
Red blood Cells |
Lymphocytes |
Mean corpuscular hemoglobin concentration (MCHC) |
Leukocytes |
Basophils |
Mean corpuscular hemoglobin (MCH) |
Eosinophils |
Mean corpuscular volume (MCV) |
Monocytes |
Red blood cell distribution width (RDW) |
Serum Glucose |
Respiratory Syncytial Virus |
Influenza A |
Influenza B |
Parainfluenza 1 |
CoronavirusNL63 |
Rhinovirus/Enterovirus |
Mycoplasma pneumoniae |
Coronavirus HKU1 |
Parainfluenza 3 |
Chlamydophila pneumoniae |
Adenovirus |
Parainfluenza 4 |
Coronavirus229E |
CoronavirusOC43 |
Inf A H1N1 2009 |
Bordetella pertussis |
Metapneumovirus |
Parainfluenza 2 |
Neutrophils |
Urea |
Proteina C reativa mg/dL |
Creatinine |
Potassium |
Sodium |
Influenza B, rapid test |
Influenza A, rapid test |
Alanine transaminase |
Aspartate transaminase |
Gamma-glutamyltransferase |
Total Bilirubin |
Direct Bilirubin |
Indirect Bilirubin |
Alkaline phosphatase |
Ionized calcium |
Strepto A |
Magnesium |
pCO2 (venous blood gas analysis) |
Hb saturation (venous blood gas analysis) |
Base excess (venous blood gas analysis) |
pO2 (venous blood gas analysis) |
Fio2 (venous blood gas analysis) |
Total CO2 (venous blood gas analysis) |
pH (venous blood gas analysis) |
HCO3 (venous blood gas analysis) |
Rods # |
Segmented |
Promyelocytes |
Metamyelocytes |
Myelocytes |
Myeloblasts |
Urine - Esterase |
Urine - Aspect |
Urine - pH |
Urine - Hemoglobin |
Urine - Bile pigments |
Urine - Ketone Bodies |
Urine - Nitrite |
Urine - Density |
Urine - Urobilinogen |
Urine - Protein |
Urine - Sugar |
Urine - Leukocytes |
Urine - Crystals |
Urine - Red blood cells |
Urine - Hyaline cylinders |
Urine - Granular cylinders |
Urine - Yeasts |
Urine - Color |
Partial thromboplastin time (PTT) |
Relationship (Patient/Normal) |
International normalized ratio (INR) |
Lactic Dehydrogenase |
Prothrombin time (PT), Activity |
Vitamin B12 |
Creatine phosphokinase (CPK) |
Ferritin |
Arterial Lactic Acid |
Lipase dosage |
D-Dimer |
Albumin |
Hb saturation (arterial blood gases) |
pCO2 (arterial blood gas analysis) |
Base excess (arterial blood gas analysis) |
pH (arterial blood gas analysis) |
Total CO2 (arterial blood gas analysis) |
HCO3 (arterial blood gas analysis) |
pO2 (arterial blood gas analysis) |
Arteiral Fio2 |
Phosphor |
ctO2 (arterial blood gas analysis) |
| 0 |
44477f75e8169d2 |
13 |
negative |
0 |
0 |
0 |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
| 1 |
126e9dd13932f68 |
17 |
negative |
0 |
0 |
0 |
0.236515 |
-0.02234 |
-0.517413 |
0.010677 |
0.102004 |
0.318366 |
-0.95079 |
-0.09461 |
-0.223767 |
-0.292269 |
1.482158 |
0.166192 |
0.357547 |
-0.625073 |
-0.140648 |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
detected |
NaN |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
-0.619086 |
1.198059 |
-0.147895 |
2.089928 |
-0.305787 |
0.862512 |
negative |
negative |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
| 2 |
a46b4402a0e5696 |
8 |
negative |
0 |
0 |
0 |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
| 3 |
f7d619a94f97c45 |
5 |
negative |
0 |
0 |
0 |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
| 4 |
d9e41465789c2b5 |
15 |
negative |
0 |
0 |
0 |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
detected |
NaN |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
a_trocar = {
'negative' : 0,
'positive' : 1
}
df['SARS-Cov-2 exam result'] = df['SARS-Cov-2 exam result'].map(a_trocar)
df.head()
|
Patient ID |
Patient age quantile |
SARS-Cov-2 exam result |
Patient addmited to regular ward (1=yes, 0=no) |
Patient addmited to semi-intensive unit (1=yes, 0=no) |
Patient addmited to intensive care unit (1=yes, 0=no) |
Hematocrit |
Hemoglobin |
Platelets |
Mean platelet volume |
Red blood Cells |
Lymphocytes |
Mean corpuscular hemoglobin concentration (MCHC) |
Leukocytes |
Basophils |
Mean corpuscular hemoglobin (MCH) |
Eosinophils |
Mean corpuscular volume (MCV) |
Monocytes |
Red blood cell distribution width (RDW) |
Serum Glucose |
Respiratory Syncytial Virus |
Influenza A |
Influenza B |
Parainfluenza 1 |
CoronavirusNL63 |
Rhinovirus/Enterovirus |
Mycoplasma pneumoniae |
Coronavirus HKU1 |
Parainfluenza 3 |
Chlamydophila pneumoniae |
Adenovirus |
Parainfluenza 4 |
Coronavirus229E |
CoronavirusOC43 |
Inf A H1N1 2009 |
Bordetella pertussis |
Metapneumovirus |
Parainfluenza 2 |
Neutrophils |
Urea |
Proteina C reativa mg/dL |
Creatinine |
Potassium |
Sodium |
Influenza B, rapid test |
Influenza A, rapid test |
Alanine transaminase |
Aspartate transaminase |
Gamma-glutamyltransferase |
Total Bilirubin |
Direct Bilirubin |
Indirect Bilirubin |
Alkaline phosphatase |
Ionized calcium |
Strepto A |
Magnesium |
pCO2 (venous blood gas analysis) |
Hb saturation (venous blood gas analysis) |
Base excess (venous blood gas analysis) |
pO2 (venous blood gas analysis) |
Fio2 (venous blood gas analysis) |
Total CO2 (venous blood gas analysis) |
pH (venous blood gas analysis) |
HCO3 (venous blood gas analysis) |
Rods # |
Segmented |
Promyelocytes |
Metamyelocytes |
Myelocytes |
Myeloblasts |
Urine - Esterase |
Urine - Aspect |
Urine - pH |
Urine - Hemoglobin |
Urine - Bile pigments |
Urine - Ketone Bodies |
Urine - Nitrite |
Urine - Density |
Urine - Urobilinogen |
Urine - Protein |
Urine - Sugar |
Urine - Leukocytes |
Urine - Crystals |
Urine - Red blood cells |
Urine - Hyaline cylinders |
Urine - Granular cylinders |
Urine - Yeasts |
Urine - Color |
Partial thromboplastin time (PTT) |
Relationship (Patient/Normal) |
International normalized ratio (INR) |
Lactic Dehydrogenase |
Prothrombin time (PT), Activity |
Vitamin B12 |
Creatine phosphokinase (CPK) |
Ferritin |
Arterial Lactic Acid |
Lipase dosage |
D-Dimer |
Albumin |
Hb saturation (arterial blood gases) |
pCO2 (arterial blood gas analysis) |
Base excess (arterial blood gas analysis) |
pH (arterial blood gas analysis) |
Total CO2 (arterial blood gas analysis) |
HCO3 (arterial blood gas analysis) |
pO2 (arterial blood gas analysis) |
Arteiral Fio2 |
Phosphor |
ctO2 (arterial blood gas analysis) |
| 0 |
44477f75e8169d2 |
13 |
0 |
0 |
0 |
0 |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
| 1 |
126e9dd13932f68 |
17 |
0 |
0 |
0 |
0 |
0.236515 |
-0.02234 |
-0.517413 |
0.010677 |
0.102004 |
0.318366 |
-0.95079 |
-0.09461 |
-0.223767 |
-0.292269 |
1.482158 |
0.166192 |
0.357547 |
-0.625073 |
-0.140648 |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
detected |
NaN |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
-0.619086 |
1.198059 |
-0.147895 |
2.089928 |
-0.305787 |
0.862512 |
negative |
negative |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
| 2 |
a46b4402a0e5696 |
8 |
0 |
0 |
0 |
0 |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
| 3 |
f7d619a94f97c45 |
5 |
0 |
0 |
0 |
0 |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
| 4 |
d9e41465789c2b5 |
15 |
0 |
0 |
0 |
0 |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
detected |
NaN |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
not_detected |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
df['SARS-Cov-2 exam result'].value_counts()
0 5086
1 558
Name: SARS-Cov-2 exam result, dtype: int64
df.replace('not_detected', 0, inplace=True)
df.replace('detected', 1, inplace = True)
df.replace('negative', 0, inplace = True)
df.replace('positive', 1, inplace = True)
df.replace('not_done', 0, inplace=True)
df = df.fillna(-1)
df.tail()
|
Patient ID |
Patient age quantile |
SARS-Cov-2 exam result |
Patient addmited to regular ward (1=yes, 0=no) |
Patient addmited to semi-intensive unit (1=yes, 0=no) |
Patient addmited to intensive care unit (1=yes, 0=no) |
Hematocrit |
Hemoglobin |
Platelets |
Mean platelet volume |
Red blood Cells |
Lymphocytes |
Mean corpuscular hemoglobin concentration (MCHC) |
Leukocytes |
Basophils |
Mean corpuscular hemoglobin (MCH) |
Eosinophils |
Mean corpuscular volume (MCV) |
Monocytes |
Red blood cell distribution width (RDW) |
Serum Glucose |
Respiratory Syncytial Virus |
Influenza A |
Influenza B |
Parainfluenza 1 |
CoronavirusNL63 |
Rhinovirus/Enterovirus |
Mycoplasma pneumoniae |
Coronavirus HKU1 |
Parainfluenza 3 |
Chlamydophila pneumoniae |
Adenovirus |
Parainfluenza 4 |
Coronavirus229E |
CoronavirusOC43 |
Inf A H1N1 2009 |
Bordetella pertussis |
Metapneumovirus |
Parainfluenza 2 |
Neutrophils |
Urea |
Proteina C reativa mg/dL |
Creatinine |
Potassium |
Sodium |
Influenza B, rapid test |
Influenza A, rapid test |
Alanine transaminase |
Aspartate transaminase |
Gamma-glutamyltransferase |
Total Bilirubin |
Direct Bilirubin |
Indirect Bilirubin |
Alkaline phosphatase |
Ionized calcium |
Strepto A |
Magnesium |
pCO2 (venous blood gas analysis) |
Hb saturation (venous blood gas analysis) |
Base excess (venous blood gas analysis) |
pO2 (venous blood gas analysis) |
Fio2 (venous blood gas analysis) |
Total CO2 (venous blood gas analysis) |
pH (venous blood gas analysis) |
HCO3 (venous blood gas analysis) |
Rods # |
Segmented |
Promyelocytes |
Metamyelocytes |
Myelocytes |
Myeloblasts |
Urine - Esterase |
Urine - Aspect |
Urine - pH |
Urine - Hemoglobin |
Urine - Bile pigments |
Urine - Ketone Bodies |
Urine - Nitrite |
Urine - Density |
Urine - Urobilinogen |
Urine - Protein |
Urine - Sugar |
Urine - Leukocytes |
Urine - Crystals |
Urine - Red blood cells |
Urine - Hyaline cylinders |
Urine - Granular cylinders |
Urine - Yeasts |
Urine - Color |
Partial thromboplastin time (PTT) |
Relationship (Patient/Normal) |
International normalized ratio (INR) |
Lactic Dehydrogenase |
Prothrombin time (PT), Activity |
Vitamin B12 |
Creatine phosphokinase (CPK) |
Ferritin |
Arterial Lactic Acid |
Lipase dosage |
D-Dimer |
Albumin |
Hb saturation (arterial blood gases) |
pCO2 (arterial blood gas analysis) |
Base excess (arterial blood gas analysis) |
pH (arterial blood gas analysis) |
Total CO2 (arterial blood gas analysis) |
HCO3 (arterial blood gas analysis) |
pO2 (arterial blood gas analysis) |
Arteiral Fio2 |
Phosphor |
ctO2 (arterial blood gas analysis) |
| 5639 |
ae66feb9e4dc3a0 |
3 |
1 |
0 |
0 |
0 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.00000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.000000 |
-1.000000 |
-1.00000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.0 |
-1.0 |
-1.00000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1 |
-1 |
-1 |
-1 |
-1 |
-1 |
-1.0 |
-1.000000 |
-1 |
-1 |
-1.0 |
-1 |
-1 |
-1.000000 |
-1 |
-1 |
-1 |
-1 |
-1.0 |
-1.0 |
-1.0 |
-1.000000 |
-1.0 |
-1.0 |
-1.000000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
| 5640 |
517c2834024f3ea |
17 |
0 |
0 |
0 |
0 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.00000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.000000 |
-1.000000 |
-1.00000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.0 |
-1.0 |
-1.00000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1 |
-1 |
-1 |
-1 |
-1 |
-1 |
-1.0 |
-1.000000 |
-1 |
-1 |
-1.0 |
-1 |
-1 |
-1.000000 |
-1 |
-1 |
-1 |
-1 |
-1.0 |
-1.0 |
-1.0 |
-1.000000 |
-1.0 |
-1.0 |
-1.000000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
| 5641 |
5c57d6037fe266d |
4 |
0 |
0 |
0 |
0 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.00000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.000000 |
-1.000000 |
-1.00000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.0 |
-1.0 |
-1.00000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1 |
-1 |
-1 |
-1 |
-1 |
-1 |
-1.0 |
-1.000000 |
-1 |
-1 |
-1.0 |
-1 |
-1 |
-1.000000 |
-1 |
-1 |
-1 |
-1 |
-1.0 |
-1.0 |
-1.0 |
-1.000000 |
-1.0 |
-1.0 |
-1.000000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
| 5642 |
c20c44766f28291 |
10 |
0 |
0 |
0 |
0 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.00000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.000000 |
-1.000000 |
-1.00000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.0 |
-1.0 |
-1.00000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.000000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1 |
clear |
5 |
absent |
absent |
absent |
-1.0 |
-0.338525 |
normal |
absent |
-1.0 |
29000 |
Ausentes |
-0.177169 |
absent |
absent |
absent |
yellow |
-1.0 |
-1.0 |
-1.0 |
-1.000000 |
-1.0 |
-1.0 |
-1.000000 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
| 5643 |
2697fdccbfeb7f7 |
19 |
1 |
0 |
0 |
0 |
0.694287 |
0.541564 |
-0.906829 |
-0.325903 |
0.578024 |
-0.295726 |
-0.353319 |
-1.288428 |
-1.140144 |
-0.135455 |
-0.835508 |
0.025985 |
0.567652 |
-0.18279 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
0.380685 |
0.453725 |
-0.50357 |
-0.735872 |
-0.552949 |
-0.934388 |
-1.0 |
-1.0 |
-0.28361 |
0.108761 |
-0.420454 |
-0.480996 |
-0.586463 |
-0.278654 |
-0.243405 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1 |
-1 |
-1 |
-1 |
-1 |
-1 |
-1.0 |
-1.000000 |
-1 |
-1 |
-1.0 |
-1 |
-1 |
-1.000000 |
-1 |
-1 |
-1 |
-1 |
-1.0 |
-1.0 |
-1.0 |
0.420204 |
-1.0 |
-1.0 |
-0.343291 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
-1.0 |
from category_encoders.ordinal import OrdinalEncoder
enc = OrdinalEncoder(cols=['Urine - Esterase', 'Urine - Aspect', 'Urine - pH', 'Urine - Hemoglobin', 'Urine - Bile pigments', 'Urine - Ketone Bodies',
'Urine - Nitrite', 'Urine - Urobilinogen', 'Urine - Leukocytes' ,'Urine - Protein', 'Urine - Sugar', 'Urine - Crystals', 'Urine - Red blood cells',
'Urine - Hyaline cylinders', 'Urine - Granular cylinders', 'Urine - Yeasts', 'Urine - Color'])
enc.fit(df)
df_ord = enc.transform(df)
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
from sklearn.metrics import recall_score
from sklearn.metrics import precision_score
x = df_ord.drop(['SARS-Cov-2 exam result','Patient ID'], axis = 1)
y = df_ord['SARS-Cov-2 exam result']
SEED = 20
treino_x, teste_x, treino_y, teste_y = train_test_split(x, y, random_state = SEED)
print('Treino com %d elementos e teste com %d elementos' % (len(treino_x), len(teste_x)))
Treino com 4233 elementos e teste com 1411 elementos
0 3816
1 417
Name: SARS-Cov-2 exam result, dtype: int64
from imblearn.over_sampling import SMOTE
smt = SMOTE()
treino_x, treino_y = smt.fit_sample(treino_x, treino_y)
array([3816, 3816], dtype=int64)
modelo = RandomForestClassifier(n_estimators=200, n_jobs=-1)
modelo.fit(treino_x, treino_y)
previsoes = modelo.predict(teste_x)
recall = recall_score(teste_y, previsoes) *100
print("O recall foi %.2f%%" % recall)
precision = precision_score(teste_y, previsoes) *100
print("A precision foi de %.2f%%" % precision)
O recall foi 58.16%
A precision foi de 13.51%
previsoes = modelo.predict_proba(teste_x)[:,1]
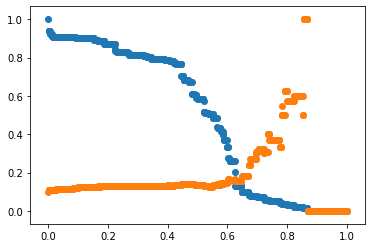
def avaliar_threshold(teste_y, previsoes):
lista_recall = []
lista_precision = []
for i in np.linspace(0,1,1000):
novas_previsoes = previsoes >= i
lista_recall.append(recall_score(teste_y, novas_previsoes))
lista_precision.append(precision_score(teste_y, novas_previsoes))
plt.scatter(np.linspace(0,1,1000), lista_recall)
plt.scatter(np.linspace(0,1,1000), lista_precision)
plt.show()
avaliar_threshold(teste_y, previsoes)
C:\Users\gust4\Anaconda3\lib\site-packages\sklearn\metrics\_classification.py:1272: UndefinedMetricWarning: Precision is ill-defined and being set to 0.0 due to no predicted samples. Use `zero_division` parameter to control this behavior.
_warn_prf(average, modifier, msg_start, len(result))